初心者セッション
データ読み込み〜データハンドリング
y__mattu
2021/3/6 TokyoR #89
はじめに
誰?
- 松村優哉
- Twitter: y__mattu
- 人材・HR Tech系で働くデータ分析屋さん
- 学生時代: 計量経済学、ベイズ統計、因果推論、マーケティング
- R歴: 7年目
- https://ymattu.github.io/
- http://y-mattu.hatenablog.com/
- Tokyo.R 運営(初心者セッションとか)
![]()
この資料の目的
- R 初心者(触ったことはあるけど、なんかよくわからない)が、雰囲気を掴む
前提知識
- Rはパッケージを使って様々な機能拡張ができる
Contents
- tidyverse について
- データの読み込み
- データハンドリング
注意
- 扱う範囲が広く資料の分量が多いので、特に重要なところをピックアップしながら進めます。
- 参考リンクも多いので資料は後でじっくり御覧ください。
- パッケージ名だけでも覚えていただけると嬉しいです。
データ分析の(おおまかな)流れ
tidyverse
tidyverse について
tidyverse(概念)
ざっくり:
- R でやるいろんな操作(データハンドリング、可視化、スクレイピング、分析、etc)を直感的で統一的なインターフェースでできるようになったら嬉しくない?
tidyverse パッケージ
- 上記の概念を実現するためのコアパッケージ群
install.packages("tidyverse")でインストール
tidyverse を読み込み
── Attaching packages ─────────────────────────────────────── tidyverse 1.3.0 ──✓ ggplot2 3.3.3 ✓ purrr 0.3.4
✓ tibble 3.1.0 ✓ dplyr 1.0.3
✓ tidyr 1.1.2 ✓ stringr 1.4.0
✓ readr 1.4.0 ✓ forcats 0.5.0── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
x dplyr::filter() masks stats::filter()
x dplyr::lag() masks stats::lag()読み込まれるパッケージ
- ggplot2: 可視化
- dplyr: データの操作
- tidyr: データを tidy に
- readr: データの読み書き
- purrr: 関数型プログラミング
- stringr: 文字列の操作
- forcats: 因子型データの操作
- tibble: tibble というモダンなデータフレーム
データの読み込み
R でのデータ読み込みのベストプラクティス
- RStudio でプロジェクトを作成
- ファイルの位置が分かりやすくなります
- 様々な読み込み関数を使って読み込み
- ローカルにあるファイル(今日の中心)
- データベース(パッケージの紹介のみ)
- Web スクレイピング(またの機会に…)
RStudio でプロジェクトを作成
Project → New Project
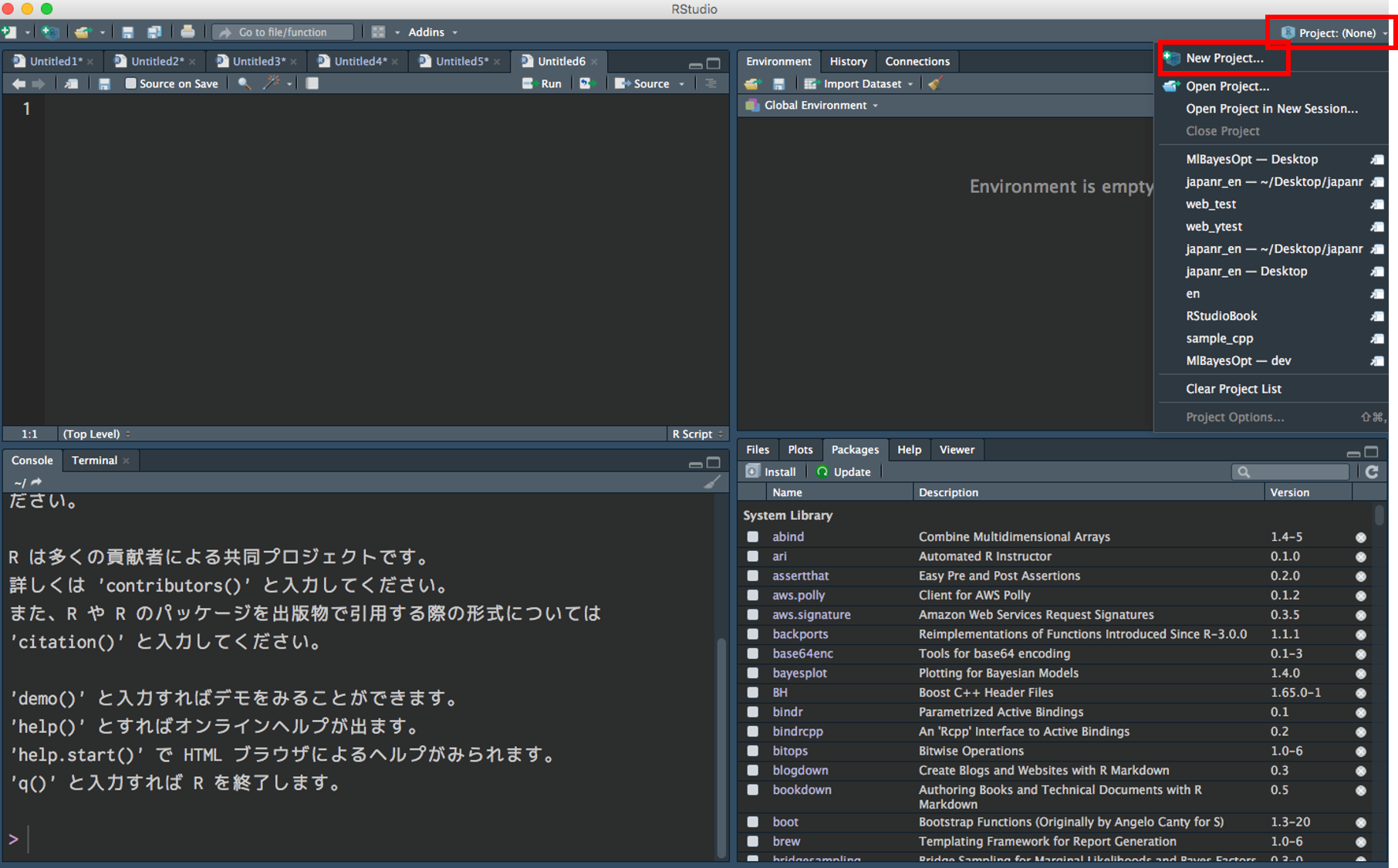
New Directory → New Project
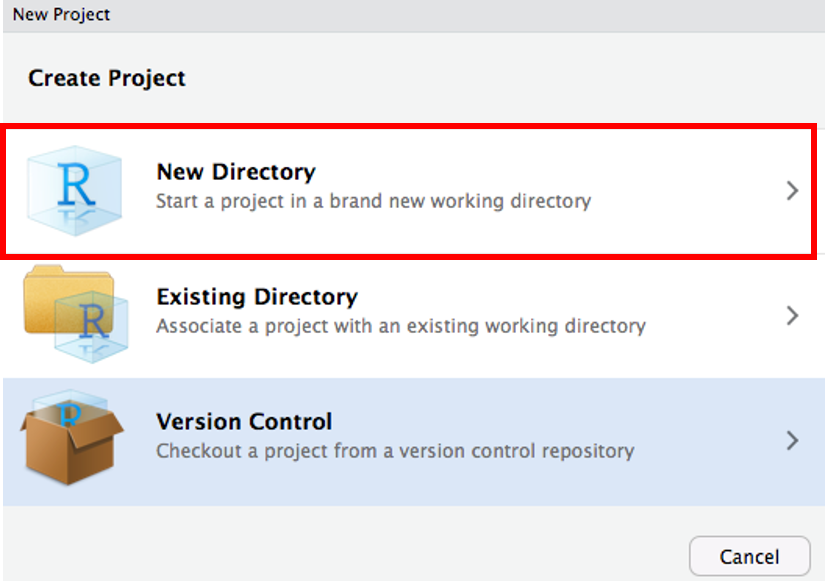

ディレクトリ名を入力
Done!
- 読み込みの関数は、プロジェクトの中のファイルを探しにいきます。
- 書籍によっては
setwd()を書いているものもありますが、RStudioプロジェクトでは必要ありません。- むしろ、RStudio プロジェクトでは非推奨
いよいよデータの読み込み
ローカルにあるファイル
csv
read.csv()
- パッケージを使わない方法
- R < 4.0.0 では
stringsAsFactors = TRUEがデフォルトになっているので、stringsAsFactors = FALSEをつけることを推奨します。
readr::read_csv()
- 高速で、列の型をいい感じにやってくれる(オススメ)
data.table::fread()
readr::read_csv()よりも高速- デフォルトでは、data.table というデータフレームとは別の形で読み込まれるのでデータフレームがいいときは
data.table = FALSE
tsv
read.delim()
read.delim()は区切り値のファイルを読む標準関数read.csv()はsep = ","をつけたもの
readr::read_tsv()
data.table::fread()
- 区切り値は勝手に判断
その他の区切り値
read.delim()
readr::read_delim()
data.table::fread()
結局?
どれがいいのか
- readrパッケージの
read_***()関数が一番オススメ - 速い、エンコーディングの調整が難しくない(後述)
| read.*** | read_*** | fread | |
|---|---|---|---|
| 速さ(45MB) | 3秒 | 0.8 秒 | 0.6秒 |
| 区切り値の判定ミス | × | × | △ |
| エンコーディング | ○ | ○ | △ |
xlsx, xls
エクセルファイル
エクセルファイルを読み込めるパッケージ
- xlsx
- gdata
- XLConnect
- openxlsx
- readxl → オススメ(速い、列の型をいい感じに読める)
読み込み方
その他の拡張子
SAS(.sas7bdat), STATA(.dta), SPSS(.sav)形式
haven パッケージで読み込み
SAS
STATA
SPSS
文字コードの指定
エンコーディング問題
- Windows の文字コードは Shift-JIS(CP932)
- Mac の文字コードは UTF8
- Windows で作られた(日本語を含む)ファイルを Mac で読むときは
Encoding=cp932 - Mac で作られた(日本語を含む)ファイルを Windows で読むときは
Encoding=UTF8
csv を CP932 で読む
R の標準関数
readr
data.table
関数とかオプション(引数)を
覚えられない
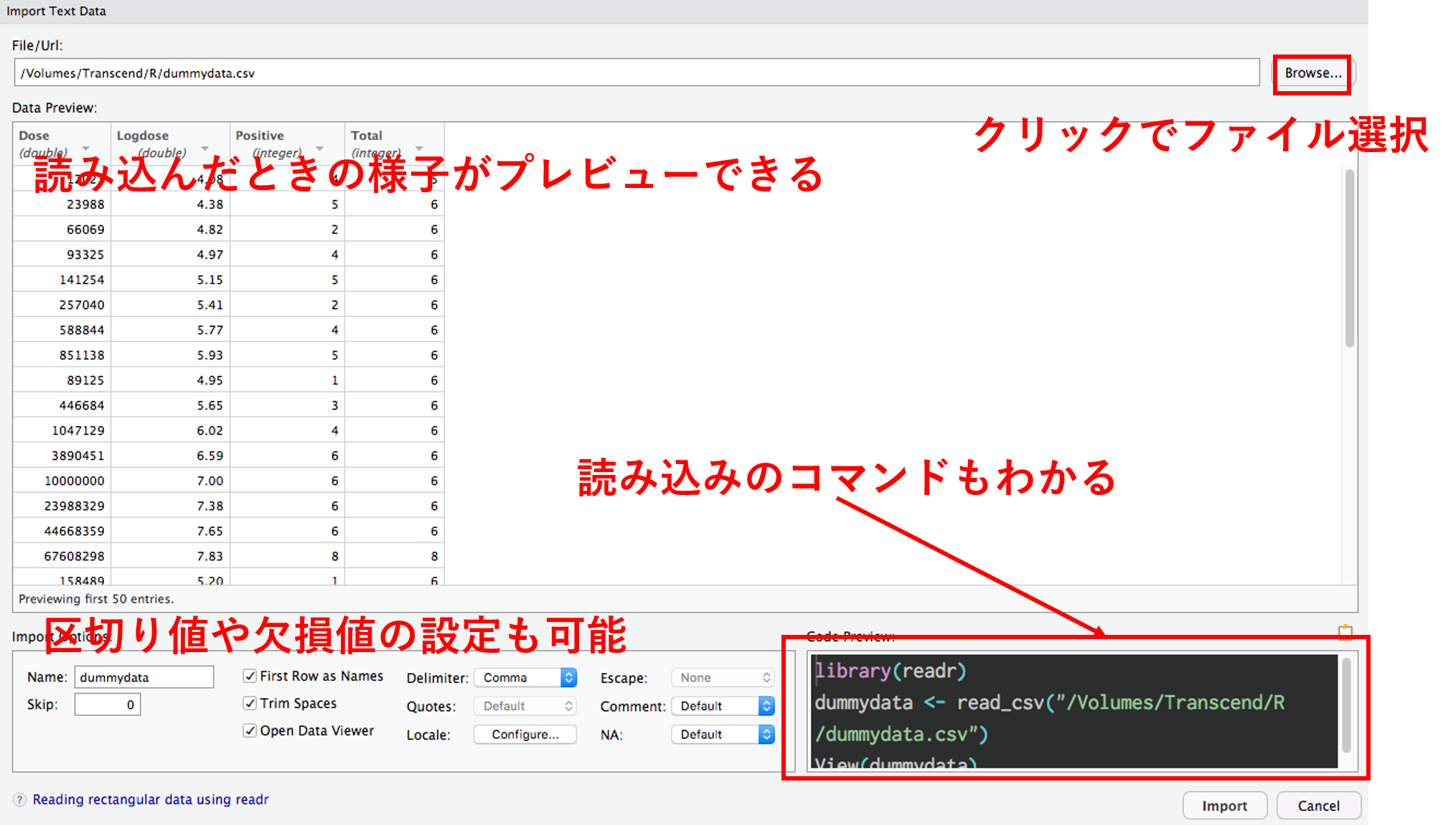
RStudio の GUI 読み込み
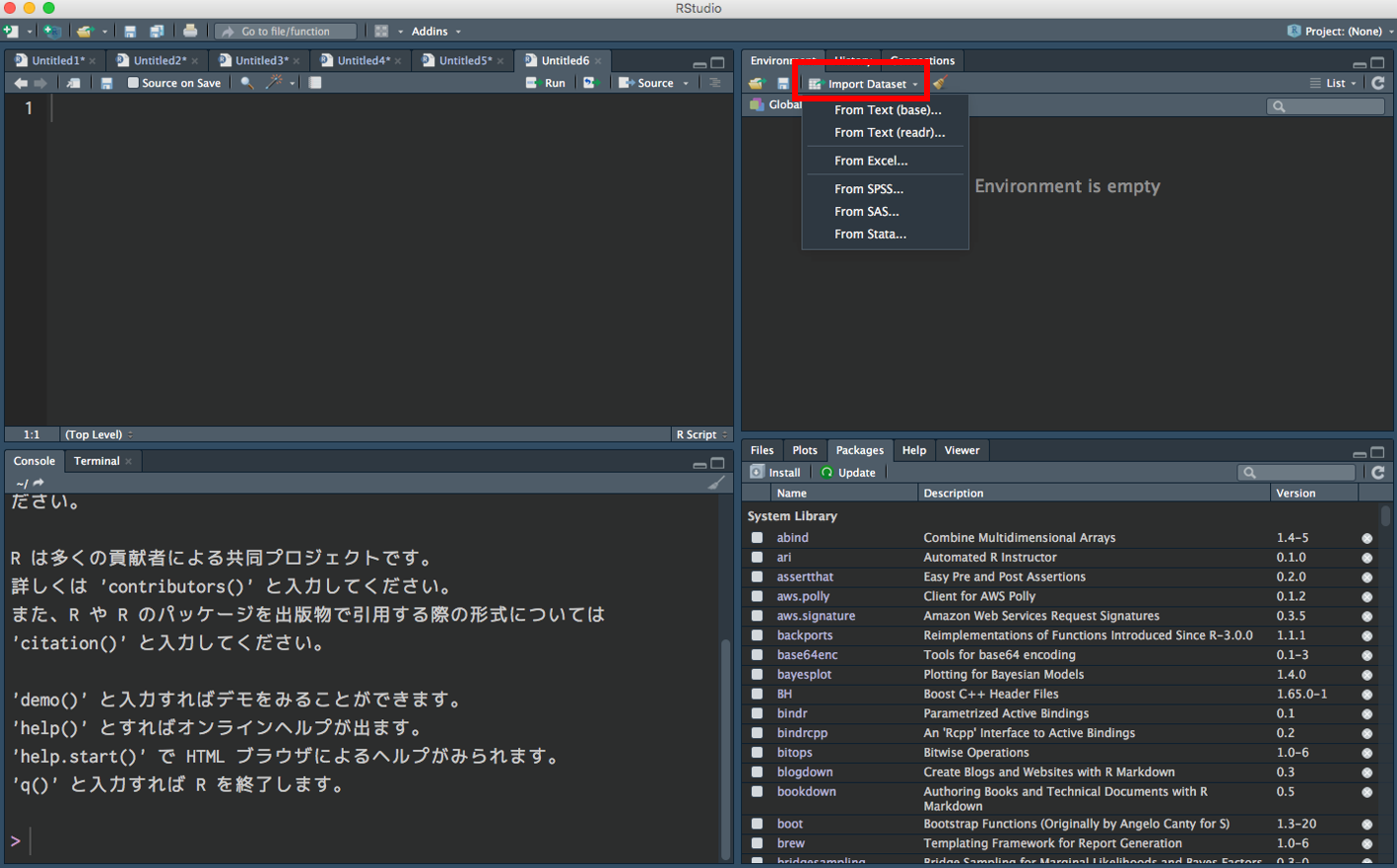
RStudio の GUI 読み込み
データハンドリング
データハンドリングでやること、例えば
- 縦横変換
- 絞り込み(列・行)
- 新しい変数の作成
- 集計
- テーブルのマージ
- etc… →分析できる形に整形
データハンドリング編のコンテンツ
- tidy data
- dplyr
- FAQ
本日の主役は


特徴
パッケージを使わないやり方より
- (大きいデータだと特に)
速い - 簡単
≒ わかりやすい - 他の tidyverse のパッケージと相性がいい
データハンドリング編のゴール
- tidy data についてざっくり理解する
- R の dplyr パッケージで簡単な集計ができるようになること
- dplyr や他のパッケージで何ができるのかをなんとなく把握して、「ググり力」を身につける
tidy data
データの形式
2つのデータ形式(例: カテゴリごとの購買金額(千円))
Wide 型
Long 型
tidy data
- 2016 年に Hadley Wickham 氏が提唱
- 定義
- 1つの列が1つの変数を表す
- 1つの行が1つの観測を表す
- 1つのテーブルが1つのデータセットを含む
- Rでのtidy data は、Long 型。
tidyr
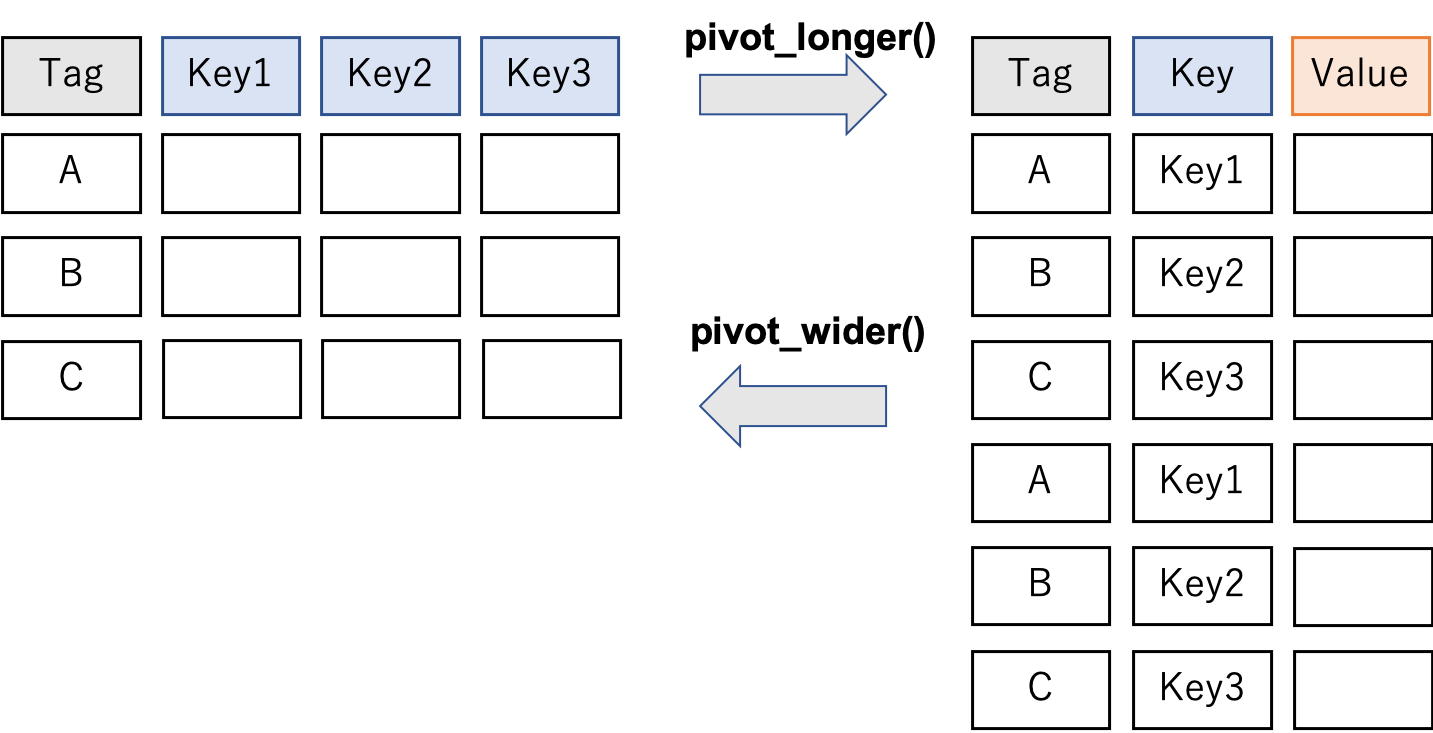
tidyrでの縦横変換の例
- 以下のデータを例に説明
- これは、いわゆる「横持ちのデータ」
df <- tibble::tibble("country" = c("a", "b", "c"),
"1999" = c(0.7, 0.3, 1.0),
"2000" = c(1.0, 2.0, 4.8),
"2001" = c(2.0, 5.0, 7.0))
df# A tibble: 3 x 4
country `1999` `2000` `2001`
<chr> <dbl> <dbl> <dbl>
1 a 0.7 1 2
2 b 0.3 2 5
3 c 1 4.8 7pivot_longer
- 横→縦(tidyな形)の変換
# A tibble: 9 x 3
country year amount
<chr> <chr> <dbl>
1 a 1999 0.7
2 a 2000 1
3 a 2001 2
4 b 1999 0.3
5 b 2000 2
6 b 2001 5
7 c 1999 1
8 c 2000 4.8
9 c 2001 7 pivot_longer
- 縦(tidyな形)→横の変換
- 統計解析のパッケージによっては、この形でないとうまく行かないものもある
# A tibble: 3 x 4
country `1999` `2000` `2001`
<chr> <dbl> <dbl> <dbl>
1 a 0.7 1 2
2 b 0.3 2 5
3 c 1 4.8 7このような場合でも
dat_m <- tibble::tibble(user = c('A', 'B', 'C'),
category_1 = c(10, 15, 8),
category_2 = c(2, 4, 5),
subject_1 = c(4, 5, 6),
subject_2 = c(5, 6, 7))
dat_m# A tibble: 3 x 5
user category_1 category_2 subject_1 subject_2
<chr> <dbl> <dbl> <dbl> <dbl>
1 A 10 2 4 5
2 B 15 4 5 6
3 C 8 5 6 7long型に変形可能
dat_long <- dat_m %>%
pivot_longer(cols = -user,
names_to = c("group", "num"),
names_sep = "_")
dat_long# A tibble: 12 x 4
user group num value
<chr> <chr> <chr> <dbl>
1 A category 1 10
2 A category 2 2
3 A subject 1 4
4 A subject 2 5
5 B category 1 15
6 B category 2 4
7 B subject 1 5
8 B subject 2 6
9 C category 1 8
10 C category 2 5
11 C subject 1 6
12 C subject 2 7詳しくは
Tokyo.R #79 の応用セッション を参照。
参考: tidyr (〜2019/09/11)
%>%
パイプ演算子
- “これまでの処理を次の関数の第 1 引数として渡す」という働き”
[1] 6例えば、以下の動作を考えてみる
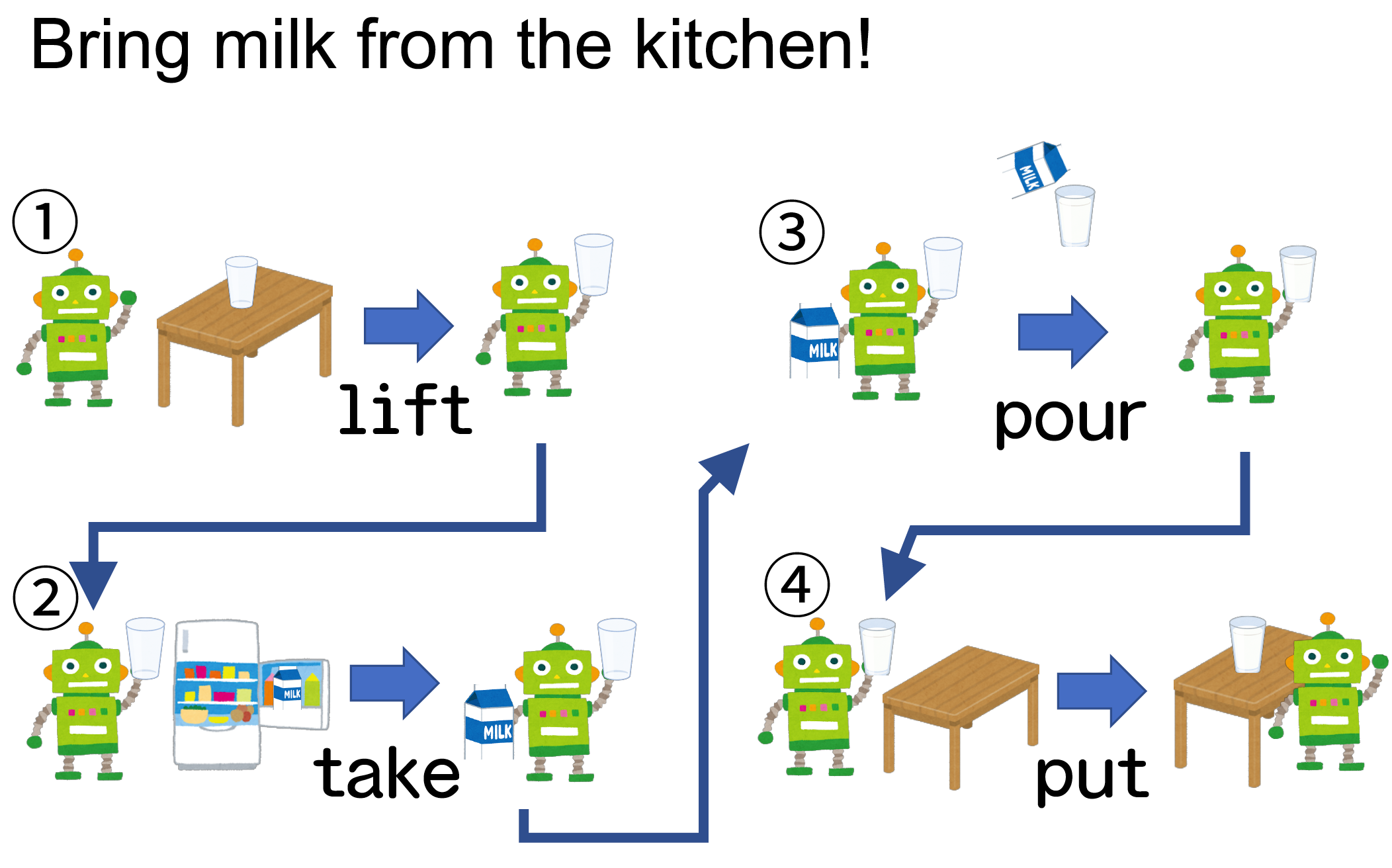
どう書くのか問題

思考の流れと書く流れ
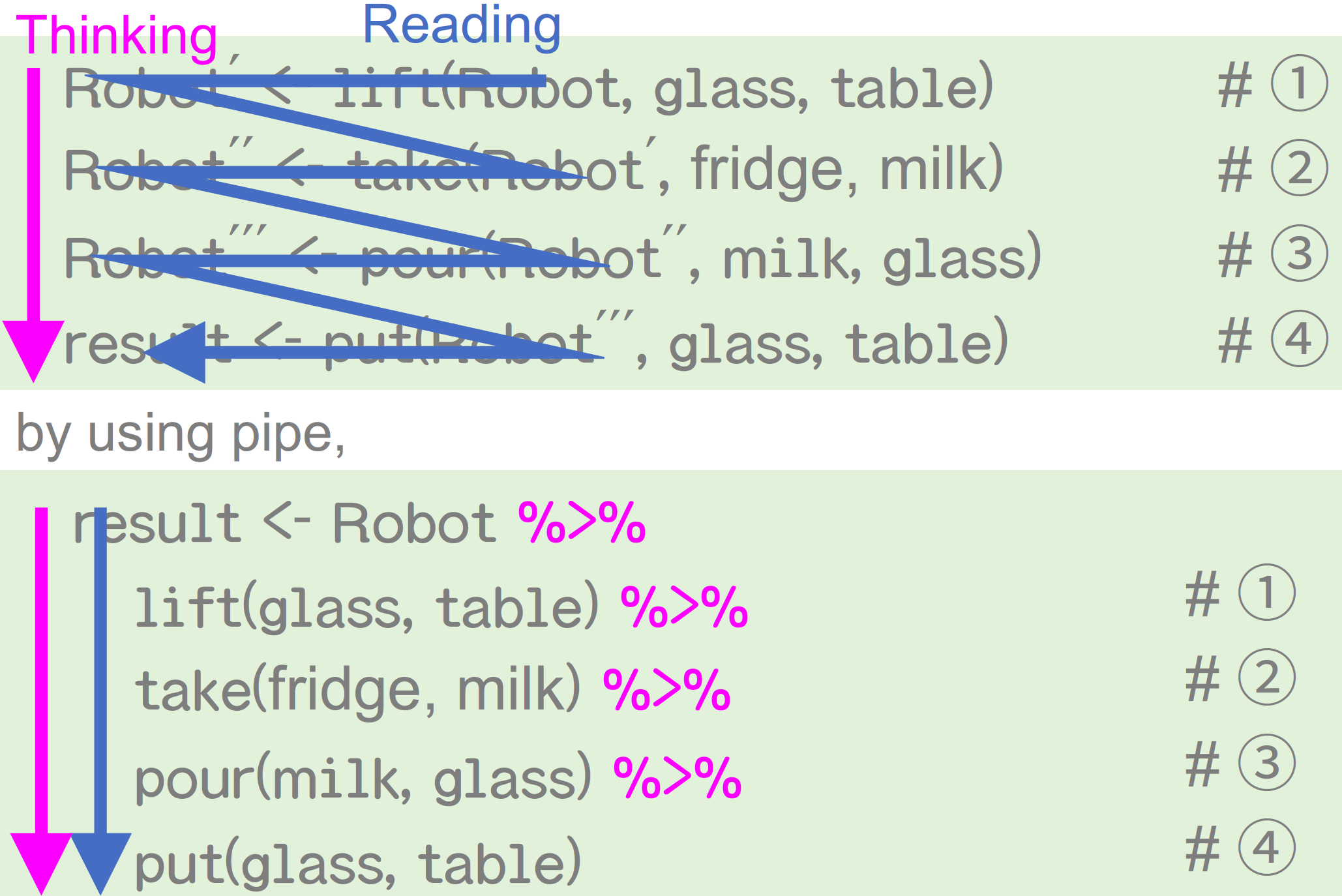
パイプ演算子を使うときのポイント
結果 <- スタート地点を書いて、やりたい処理をパイプでつないでいく- RStudioでのキーボードショートカット
- Windows:
Ctrl+Shift+M - Mac:
Cmd+Shift+M
- Windows:
扱うデータ
EC サイトのログデータ
- を意識して作ったダミーデータ
- https://github.com/ymattu/sampledata_small
![データの説明]()
データの読み込み方
- RStudio のプロジェクトを作成
- Terminal ペインで以下を実行
git clone https://github.com/ymattu/sampledata_small- readr パッケージの関数で読み込み
dplyr のデータハンドリング基礎
列選択
列選択のやりかたいろいろ 2
例
select(product, 1:3) # 列番号が連続している場合
select(product, ProductID:Price) # 列名でも連続していれば同様
select(product, -CreatedDate) # 特定の列を除く
select(product, -4) # 特定の列番号を除く
select(product, starts_with("p")) # "p"で始まる列のみを選択
select(product, starts_with("p"), ignore.case = TRUE) # 大文字小文字を無視
select(product, matches("^(Product|Price)")) # "Product"または"Price"で始まる列を選択列追加
- 税込み価格を計算
行の絞り込み
集計
- グルーピング + 集計
ここまでやったところで
パッケージを使わないでできないの?
- できるものもあります。
- select, filter あたりはできます。
- でもめんどくさい
- しかもデータが大きいと遅い
- このあたり、私の過去資料もみてね
- でも
$はお手軽だしよく使います。
$で 1 列だけ取り出す
[1] "雑貨・日用品" "花・グリーン" "食品"
[4] "衣料品" "ヘルス&ビューティー" "家具・インテリア・家電"日付の操作
lubridate パッケージ

データハンドリングでの使い所
たくさんあるけど例えば
ここから集計につなげる
ユーザー、年ごとに集計
その他、代表的な
(面倒くさい)型たち
文字列型
- stringr パッケージ
- https://kazutan.github.io/kazutanR/stringr-intro.html
因子型(factor 型)
- forcats パッケージ
- https://kazutan.github.io/kazutanR/forcats_test.html
テーブルのマージ
複数のテーブルを考える
a
b
- 基本は SQL と同じ
inner_join()
a
b
left_join()
a
b
full_join()
a
b
anti_join()
a
b
FAQ
dplyr とかだと何で
R の標準関数より速いの?
Answer : C++を使っているから
- dplyrや readrでは、メインの処理を C++でやり、結果を R で受け取る、という構造になっています。
- Rcpp パッケージが活躍!
たくさんのテーブルを join したい!
例えばこんな感じ(a, b, c 3 つのデータ)
x1 x2
1 A 1
2 B 2
3 C 3 x1 x3
1 A TRUE
2 B FALSE
3 D TRUE x1 x4
1 B 10
2 C 11
3 D 12たくさんのテーブルを join したい!
Answer : 少し応用的ですが、purrrパッケージを使うと簡単です。
purrr パッケージの参考資料→そろそろ手を出す purrr
dplyr1.0.0と
いくつかの関数の変化について
dplyr 1.0.0
- 2020/6/22に、dplyrパッケージのバージョンが 0.X.Xから 1.0.0 へバージョンアップした
- ソフトウェアの世界では、このようなメジャーアップデートは大きな意味を持つ
- dplyrでも、いくつかの破壊的な変更があった
- 古いバージョンで分析結果が再現しなくなる可能性があるため、要注意
- 以下の内容を紹介
summarise()の挙動変化rowwise()関数について- 追加関数(
across(),where())について
例示のデータ
- starwarsデータ(dplyrパッケージに含まれる)
Rows: 87
Columns: 14
$ name <chr> "Luke Skywalker", "C-3PO", "R2-D2", "Darth Vader", "Leia Or…
$ height <int> 172, 167, 96, 202, 150, 178, 165, 97, 183, 182, 188, 180, 2…
$ mass <dbl> 77.0, 75.0, 32.0, 136.0, 49.0, 120.0, 75.0, 32.0, 84.0, 77.…
$ hair_color <chr> "blond", NA, NA, "none", "brown", "brown, grey", "brown", N…
$ skin_color <chr> "fair", "gold", "white, blue", "white", "light", "light", "…
$ eye_color <chr> "blue", "yellow", "red", "yellow", "brown", "blue", "blue",…
$ birth_year <dbl> 19.0, 112.0, 33.0, 41.9, 19.0, 52.0, 47.0, NA, 24.0, 57.0, …
$ sex <chr> "male", "none", "none", "male", "female", "male", "female",…
$ gender <chr> "masculine", "masculine", "masculine", "masculine", "femini…
$ homeworld <chr> "Tatooine", "Tatooine", "Naboo", "Tatooine", "Alderaan", "T…
$ species <chr> "Human", "Droid", "Droid", "Human", "Human", "Human", "Huma…
$ films <list> [<"The Empire Strikes Back", "Revenge of the Sith", "Retur…
$ vehicles <list> [<"Snowspeeder", "Imperial Speeder Bike">, <>, <>, <>, "Im…
$ starships <list> [<"X-wing", "Imperial shuttle">, <>, <>, "TIE Advanced x1"…summarise()の挙動変化について
そもそも summarise() のはたらきは?
- その1: グループ化されたデータフレームの値を集約し、グループ化を1つ解除する
# A tibble: 6 x 14
# Groups: species, homeworld [4]
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
4 Darth V… 202 136 none white yellow 41.9 male mascu…
5 Leia Or… 150 49 brown light brown 19 fema… femin…
6 Owen La… 178 120 brown, grey light blue 52 male mascu…
# … with 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>集約してみる
starwars_summary1 <- grouped_starwars %>%
summarise(mean_height = mean(height, na.rm = TRUE))
starwars_summary1# A tibble: 58 x 3
# Groups: species [38]
species homeworld mean_height
<chr> <chr> <dbl>
1 Aleena Aleen Minor 79
2 Besalisk Ojom 198
3 Cerean Cerea 198
4 Chagrian Champala 196
5 Clawdite Zolan 168
6 Droid Naboo 96
7 Droid Tatooine 132
8 Droid <NA> 148
9 Dug Malastare 112
10 Ewok Endor 88
11 Geonosian Geonosis 183
12 Gungan Naboo 209.
13 Human Alderaan 176.
14 Human Bespin 175
15 Human Bestine IV 180
16 Human Chandrila 150
17 Human Concord Dawn 183
18 Human Corellia 175
19 Human Coruscant 168.
20 Human Eriadu 180
21 Human Haruun Kal 188
22 Human Kamino 183
23 Human Naboo 168.
24 Human Serenno 193
25 Human Socorro 177
26 Human Stewjon 182
27 Human Tatooine 179.
28 Human <NA> 193
29 Hutt Nal Hutta 175
30 Iktotchi Iktotch 188
31 Kaleesh Kalee 216
32 Kaminoan Kamino 221
33 Kel Dor Dorin 188
34 Mirialan Mirial 168
35 Mon Calamari Mon Cala 180
36 Muun Muunilinst 191
37 Nautolan Glee Anselm 196
38 Neimodian Cato Neimoidia 191
39 Pau'an Utapau 206
40 Quermian Quermia 264
41 Rodian Rodia 173
42 Skakoan Skako 193
43 Sullustan Sullust 160
44 Tholothian Coruscant 184
45 Togruta Shili 178
46 Toong Tund 163
47 Toydarian Toydaria 137
48 Trandoshan Trandosha 190
49 Twi'lek Ryloth 179
50 Vulptereen Vulpter 94
51 Wookiee Kashyyyk 231
52 Xexto Troiken 122
53 Yoda's species <NA> 66
54 Zabrak Dathomir 175
55 Zabrak Iridonia 171
56 <NA> Naboo 183
57 <NA> Umbara 178
58 <NA> <NA> NaN グループ化が残っていると不都合なこと
- 例えば、集計された身長が 登場人物全員 の平均と比べてどうなのかを見てみたい(平均の平均を取るのが適切でないという議論はさておき)
- ここでの
mean()では、グループ化された中での平均を計算してしまう
# A tibble: 58 x 4
# Groups: species [38]
species homeworld mean_height rel_heigt
<chr> <chr> <dbl> <dbl>
1 Aleena Aleen Minor 79 1
2 Besalisk Ojom 198 1
3 Cerean Cerea 198 1
4 Chagrian Champala 196 1
5 Clawdite Zolan 168 1
6 Droid Naboo 96 0.766
7 Droid Tatooine 132 1.05
8 Droid <NA> 148 1.18
9 Dug Malastare 112 1
10 Ewok Endor 88 1
11 Geonosian Geonosis 183 1
12 Gungan Naboo 209. 1
13 Human Alderaan 176. 0.989
14 Human Bespin 175 0.982
15 Human Bestine IV 180 1.01
16 Human Chandrila 150 0.842
17 Human Concord Dawn 183 1.03
18 Human Corellia 175 0.982
19 Human Coruscant 168. 0.945
20 Human Eriadu 180 1.01
21 Human Haruun Kal 188 1.05
22 Human Kamino 183 1.03
23 Human Naboo 168. 0.945
24 Human Serenno 193 1.08
25 Human Socorro 177 0.993
26 Human Stewjon 182 1.02
27 Human Tatooine 179. 1.01
28 Human <NA> 193 1.08
29 Hutt Nal Hutta 175 1
30 Iktotchi Iktotch 188 1
31 Kaleesh Kalee 216 1
32 Kaminoan Kamino 221 1
33 Kel Dor Dorin 188 1
34 Mirialan Mirial 168 1
35 Mon Calamari Mon Cala 180 1
36 Muun Muunilinst 191 1
37 Nautolan Glee Anselm 196 1
38 Neimodian Cato Neimoidia 191 1
39 Pau'an Utapau 206 1
40 Quermian Quermia 264 1
41 Rodian Rodia 173 1
42 Skakoan Skako 193 1
43 Sullustan Sullust 160 1
44 Tholothian Coruscant 184 1
45 Togruta Shili 178 1
46 Toong Tund 163 1
47 Toydarian Toydaria 137 1
48 Trandoshan Trandosha 190 1
49 Twi'lek Ryloth 179 1
50 Vulptereen Vulpter 94 1
51 Wookiee Kashyyyk 231 1
52 Xexto Troiken 122 1
53 Yoda's species <NA> 66 1
54 Zabrak Dathomir 175 1.01
55 Zabrak Iridonia 171 0.988
56 <NA> Naboo 183 NaN
57 <NA> Umbara 178 NaN
58 <NA> <NA> NaN NaN ungroup() されたのが正しい結果
# A tibble: 58 x 4
species homeworld mean_height rel_heigt
<chr> <chr> <dbl> <dbl>
1 Aleena Aleen Minor 79 NaN
2 Besalisk Ojom 198 NaN
3 Cerean Cerea 198 NaN
4 Chagrian Champala 196 NaN
5 Clawdite Zolan 168 NaN
6 Droid Naboo 96 NaN
7 Droid Tatooine 132 NaN
8 Droid <NA> 148 NaN
9 Dug Malastare 112 NaN
10 Ewok Endor 88 NaN
11 Geonosian Geonosis 183 NaN
12 Gungan Naboo 209. NaN
13 Human Alderaan 176. NaN
14 Human Bespin 175 NaN
15 Human Bestine IV 180 NaN
16 Human Chandrila 150 NaN
17 Human Concord Dawn 183 NaN
18 Human Corellia 175 NaN
19 Human Coruscant 168. NaN
20 Human Eriadu 180 NaN
21 Human Haruun Kal 188 NaN
22 Human Kamino 183 NaN
23 Human Naboo 168. NaN
24 Human Serenno 193 NaN
25 Human Socorro 177 NaN
26 Human Stewjon 182 NaN
27 Human Tatooine 179. NaN
28 Human <NA> 193 NaN
29 Hutt Nal Hutta 175 NaN
30 Iktotchi Iktotch 188 NaN
31 Kaleesh Kalee 216 NaN
32 Kaminoan Kamino 221 NaN
33 Kel Dor Dorin 188 NaN
34 Mirialan Mirial 168 NaN
35 Mon Calamari Mon Cala 180 NaN
36 Muun Muunilinst 191 NaN
37 Nautolan Glee Anselm 196 NaN
38 Neimodian Cato Neimoidia 191 NaN
39 Pau'an Utapau 206 NaN
40 Quermian Quermia 264 NaN
41 Rodian Rodia 173 NaN
42 Skakoan Skako 193 NaN
43 Sullustan Sullust 160 NaN
44 Tholothian Coruscant 184 NaN
45 Togruta Shili 178 NaN
46 Toong Tund 163 NaN
47 Toydarian Toydaria 137 NaN
48 Trandoshan Trandosha 190 NaN
49 Twi'lek Ryloth 179 NaN
50 Vulptereen Vulpter 94 NaN
51 Wookiee Kashyyyk 231 NaN
52 Xexto Troiken 122 NaN
53 Yoda's species <NA> 66 NaN
54 Zabrak Dathomir 175 NaN
55 Zabrak Iridonia 171 NaN
56 <NA> Naboo 183 NaN
57 <NA> Umbara 178 NaN
58 <NA> <NA> NaN NaNそもそも summarise() のはたらきは?
- その2: 集約された結果は複数の値になってはいけない
grouped_starwars %>%
summarise(q_height = quantile(height, na.rm = TRUE))
#> Error: Column `dep_height` must be length 1 (a summary value), not 5参考
quantile() は分位数を返す集約関数だが、出力は5つ
0% 25% 50% 75% 100%
66 167 180 191 264 summarise() の新機能1
- 親切なメッセージ
- この場合、挙動は変わらない
summarise() の新機能2
- .groups 引数
- “drop_last”: 一番うしろのグループ化変数を解除。これまでと同じ挙動で、dplyr1.0.0でも基本は(後述)これがデフォルト。
- “drop”: すべてのグループ化を解除。
- “keep”: グループ化をそのまま維持。
- “rowwise”: rowwise な状態に(後述)。
- 挙動については、デモで。
summarise() の新機能3
- 集約後の値が複数でも大丈夫
- 集約値が複数の場合、.groups引数は自動的に “keep” になる
grouped_starwars %>%
summarise(q_height = quantile(height, na.rm = TRUE))
# A tibble: 290 x 3
# Groups: species, homeworld [58]
# species homeworld dep_height
# <chr> <chr> <dbl>
# 1 Aleena Aleen Minor 79
# 2 Aleena Aleen Minor 79
# 3 Aleena Aleen Minor 79
# 4 Aleena Aleen Minor 79
# 5 Aleena Aleen Minor 79
# 6 Besalisk Ojom 198
# 7 Besalisk Ojom 198
# 8 Besalisk Ojom 198
# 9 Besalisk Ojom 198
# 10 Besalisk Ojom 198
# … with 280 more rowsどの分位点か、わからないので…
grouped_starwars %>%
summarise(q = c(0, 0.25, 0.5, 0.75, 1),
q_height = quantile(height, na.rm = TRUE))
# A tibble: 290 x 4
# Groups: species, homeworld [58]
# species homeworld q q_height
# <chr> <chr> <dbl> <dbl>
# 1 Aleena Aleen Minor 0 79
# 2 Aleena Aleen Minor 0.25 79
# 3 Aleena Aleen Minor 0.5 79
# 4 Aleena Aleen Minor 0.75 79
# 5 Aleena Aleen Minor 1 79
# 6 Besalisk Ojom 0 198
# 7 Besalisk Ojom 0.25 198
# 8 Besalisk Ojom 0.5 198
# 9 Besalisk Ojom 0.75 198
# 10 Besalisk Ojom 1 198
# … with 280 more rowsacross(), where()について
複数の列に、同じ処理をしたい場合1
- dplyr < 1.0.0 における
***_at()の例- speciesでグルーピングして、「gender, homeworld」についてユニークな値を数える場合
starwars %>%
group_by(species) %>%
filter(n() > 1) %>%
summarise_at(vars(gender, homeworld),
n_distinct)# A tibble: 9 x 3
species gender homeworld
<chr> <int> <int>
1 Droid 2 3
2 Gungan 1 1
3 Human 2 16
4 Kaminoan 2 1
5 Mirialan 1 1
6 Twi'lek 2 1
7 Wookiee 1 1
8 Zabrak 1 2
9 <NA> 1 3複数の列に、同じ処理をしたい場合2
***_at()の他に、- 「すべての列に対して同じ処理を行う」場合→
***_all() - 「特定の方の列(例: 数値型)に対して同じ処理を行う」場合→
***_if()
- 「すべての列に対して同じ処理を行う」場合→
- *** には、summarise, mutateが使える
dplyr >= 1.0.0 で推奨される書き方
across(),where()を使用- あくまで「推奨」であって、
***_at(), `***_aif(),***_all()がなくなるわけではない
| 対象の列 | dplyr < 1.0.0 | dplyr >= 1.0.0 |
|---|---|---|
| 列名を指定 | ***_at | ***(across(列の指定, 処理)) |
| 特定の型を持つ列 | ***_if | ***(across(where(列の型の指定), 処理)) |
| 全ての列 | ***_all | ***(across(everything(), 処理)) |
rowwise() について
そもそもどういう場面で使う関数か?
- dplyrで使われる関数は、ベクトルの処理を基本としている
- つまり、列方向の操作が基本
- 逆にレコード方向(横方向)の操作は苦手
- 以下のデータを例に説明
x y
1 1 3
2 2 3
3 3 3
4 1 2
5 2 2
6 3 2
7 1 1
8 2 1
9 3 1dplyrはレコード方向(横方向)の操作は苦手
- xとyの平均を取りたいが、なにかおかしい
- この場合、1行目のxとyの変換がすべてに適用されてしまっている
x y m
1 1 3 2
2 2 3 2
3 3 3 2
4 1 2 2
5 2 2 2
6 3 2 2
7 1 1 2
8 2 1 2
9 3 1 2rowwise()でこれを解消
- rowwiseは「1行1グループ」に変換する関数
- グループ内で計算をするため、うまくいく
# A tibble: 9 x 3
# Rowwise:
x y m
<int> <int> <dbl>
1 1 3 2
2 2 3 2.5
3 3 3 3
4 1 2 1.5
5 2 2 2
6 3 2 2.5
7 1 1 1
8 2 1 1.5
9 3 1 2 rowwise()の歴史
- 2014年頃(dplyr0.2くらい)
→rowwise()はすでにあった - 2017年頃(dplyr0.7くらい)
→purrrパッケージやtidyr::nest()の充実で推奨されなくなる (将来的になくなるとの見解も) - 2019年頃(dplyr0.8くらい)
→questioning(使ってもいいけど、ちょっとどうなんだろう?でも代替手段はない)というステータスになっていた - 2020年(dply 1.0.0)
→正式に使ってOKな関数に
まとめ
Contents
- R と RStudio について
- データハンドリング編
- tidyverse について
- readrパッケージを用いたテーブルデータの読み込み
- dplyr, tidyrパッケージを用いたデータハンドリング
